遺伝疫学におけるデータ・クリーニング

遺伝疫学では遺伝情報と疾患の関連性を見ることが多いですが、データそのものにエラーが隠れていることもあります。
このため、集めたデータにerrorがないかをいくつかの方法を使用して確認します。
遺伝情報にはある程度のパターンがあるため(メンデルの法則など)、これらを利用してデータの質が担保されているのかを確認する作業が必要となります。
遺伝疫学について詳しく学びたい方は以下の本がオススメです↓
遺伝疫学において、データのエラーを見つける
遺伝疫学では、遺伝情報はgenotypingを用いて集めますが、error(誤り)が起こることがあります。ErrorにもFalse positive (偽陽性)とFalse negative (偽陰性)があります。
このErrorを確認する方法はいくつかありますが、特にSNP genotypeは、Mendelian checkのみではすり抜けてしまうことがあるので、注意が必要です。
まずは、代表的なMendelican Checkについて解説していきましょう。
Mendelian Checkについて
Mendelian Checkですが、その名の通りメンデルの法則に従っているかに基づいて確認をしていく方法です。
まずは以下の例を見てみましょう。
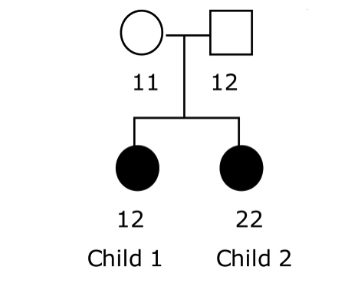
Mendelian Checkの例
父の遺伝情報が1/2、母の遺伝情報が1/1であったとします。
この場合、子供の遺伝情報が、1/1 or 1/2 になるはずですが、2/2の子供がいる場合、Mendelian checkでデータがおかしい点を指摘することができます。
Mendelian (genetic) Inconsistencyについて
以下の3つに当てはまる場合もメンデルの法則に従っていない可能性があります:
- 同胞内で5つ以上のalleleがある
- 同胞内でhomozygousの子供がいるのに、4つ以上のalleleがある
- 同胞内で異なるhomozygousの子供が2人いるのに、3つ以上のalleleがある
少しイメージしづらいかもしれないので、実例を見ながら考えていきましょう。
1:同胞内で5つ以上のalleleがある
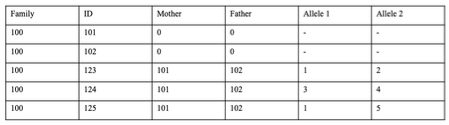
この例では、父・母の遺伝情報は不明ですが、子供三人のalleleの情報が分かっていたとします。三人の子供の遺伝情報は以下の通りでした
- ID123: 1/2
- ID124: 3/4
- ID125: 1/5
ID123とID124の二人の遺伝情報から、父と母は、1/3と2/4のalleleの組み合わせであると予測できます。
しかし、ID125の子供を見ると、1/5の遺伝情報を有しており、矛盾するのが分かるでしょう。
2:同胞内でhomozygousの子供がいるのに、4つ以上のalleleがある
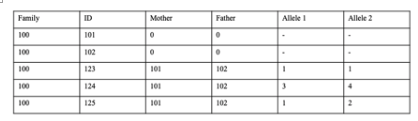
同様に、父・母の遺伝情報がわからない状況で、ID123の子供のAlleleが1/1であったとします。この場合、父・母ともに1のalleleを持っているはずで、残りの2つが異なるalleleであったとしても(例: 3と4)、alleleは合計で3つまでです。
この場合、ID124は矛盾しませんが、ID125の子供が矛盾することになります。
3:同胞内で異なるhomozygousの子供が2人いるのに、3つ以上のalleleがある
同胞内で異なるhomozygousの子供が2人いるのに、3つ以上のalleleがある
こちらのケースでは、ID123は1/1、ID124は2/2と分かっています。
この場合、父・母のAlleleはともに1/2であり、子供の組み合わせとしては1/1、2/2、1/2のみです。
ですが、ID125の子供が1/3のalleleを有しており、メンデルの法則に矛盾しているのがわかります。
データファイル上の問題
遺伝情報はデータとしてファイルに保存をしますが、様々な問題でエラーが起こることがあります。よくある問題として、
- 保存するファイルのformatが間違っていた
- 欠損値があった(年齢・性別など)
- 性別が間違っていた
- 範囲外のalleleであった
などが該当します。
SNP Genotypingの問題
SNPのgenotypingは、一般的にはとても正確です。
このため、errorは「問題のあるSNPs」に集簇する傾向があります。
また、errorを放置して最終解析に向かった場合、バイアス混じりの結果を報告してしまう可能性があります。
このため、最終的な解析をする前にerrorを特定して除外しておく必要があります。
質の悪いデータとは?
質の悪いデータとは、
- genotypingのエラーが多い
- SNPsがよくない
- サンプルがよくない
といった問題に由来します。
質の低いデータを探す方法
質の低いデータを探す方法ですが、一般的には
- SNPsのデータやサンプル間での違いの統計を確認する
- コントロールを解析に入れて一致しているか確認する
- HapMap(a Haplotype map)をコントロールとして、比較する
などがあります。
しかし、例えばHapmapにもエラーがあることがあり、少し注意が必要なこともあります。
質の低いSNPsを見つける方法
質の低いSNPsを見つける方法ですが、
- Call rate
- メンデルの法則
- Hardy Weinbergの法則
- Quality score
といった指標を使用して、質をチェックします。
しかし、これらを利用して質をチェックしたとしても、通過してしまうことがあります。
質の低いサンプルを見つける方法
SNPsなどのgenotypeのプロセス問題がなくても、サンプル自体の問題があって間違って評価されてしまうケースもあります。
サンプルレベルのデータから、call rateを確認する作業をします。
サンプルが悪い場合は修正不可能なこともあり、最終的な解析から除外する必要がある時もあります。
遺伝疫学について詳しく学びたい方は以下の本がオススメです↓