連鎖解析(linkage analysis)、LODスコア、Recombination fractionについて [遺伝疫学]

今回は、Genetic linkage(遺伝的連鎖)の解析で必要な背景知識と、遺伝疫学で使用しているモデルとの関連を解説していこうと思います。
具体的には、
- Recombination fraction (θ)
- Linkage analyses(連鎖解析)
- LOD score(LODスコア)
の方法論について実用例を交えながら解説していきます。
遺伝的連鎖(genetic linkage)について詳しく知りたい方は、前回の記事をご参照ください。

また、遺伝疫学について一通りの学習をしたい方は、以下の教科書をお勧めしています。
Genetic Linkageとは?
Genetic linkage(遺伝的連鎖)とは、特定の対立遺伝子の組み合わせが、メンデルの独立の法則に従わずに、親から子へ一緒に遺伝する遺伝学的現象のことを言います。
このgenetic linkageと遺伝疫学を結びつけて考える前に、まずは配偶子の組み換え(recombination)について理解しましょう。
Centimorgan (cM)について
前回、genetic linkage(遺伝的連鎖)の解説をしました。同一染色体の場合、遺伝子同士が近ければrecombination(組み換え)は起こりづらく、遠ければ起こる確率が高まる点を解説してきました。

この現象を逆から考えると、recombinationが起こる確率の高い遺伝子同士は染色体上で遠い位置に、確率が低い場合は近いい位置にいるのがわかります。
これを利用してDNAの距離を定義したのがCentimorgan (cM)となります。
Recombination fraction:θ(theta)
次に、Recombination fraction (θ)について解説していきましょう。
θは2つの遺伝子座(loci)でrecombinationが生じる確率のことを言います。
計算はシンプルでして、
- θ= #Recombinants /(#Recombinants + #non-recombinants)
となります。
θも遺伝子間の距離になるため、値が小さければ近く、大きければ遠くなります。
イメージとしては以下のようになります。
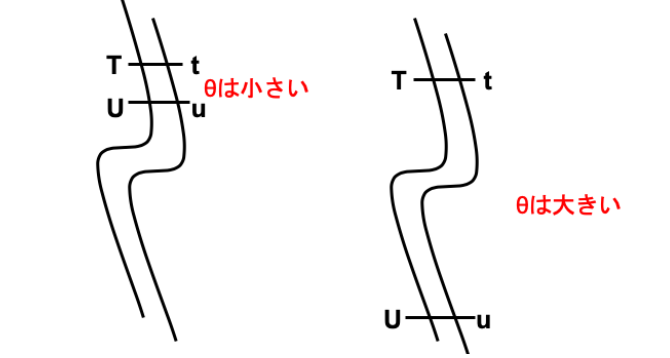
θは0〜0.5の間の値をとります。
例1:θを計算する
以下の例で考えてみましょう。
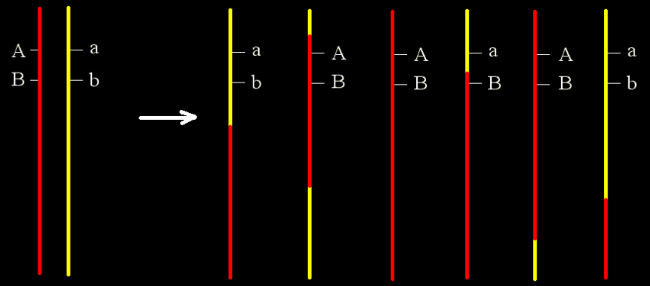
2つのpaternal alleleから6つのalleleが生じたと考えましょう。
θはいくつになるでしょうか?
右の6本の染色体でrecombinantはaBのみです。よって#recombinants = 1となります。
また、#non-recombinants = 6 – 1 = 5となります。よってθは、
- θ= 1 / (1 + 5) = 1/6 = 0.17
となります。
例2:θの計算
Nail-patella syndrome (NPS)は日本語で爪膝蓋症候群という疾患ですが、原因遺伝子がABP血液型の遺伝子に比較的近い場所にあるのが知られています。
この性質を利用して、θを計算してみましょう。
まず、以下の図を見てください。
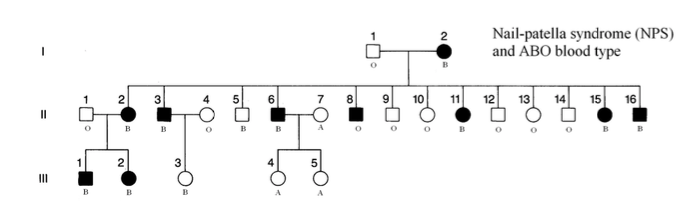
●や■がNPSを発症したことを示していますが、B型の場合、NPSを起こす可能性が高いことに気がつくと思います。
ここで、ABO血液型の遺伝型を以下のように定義します:
- A型= IAIAor IAi
- B型= IBIBor IBi
- AB型= IAIB
- O型= ii
ABO型をgenotypeに変換すると上の図は以下のようになりました:
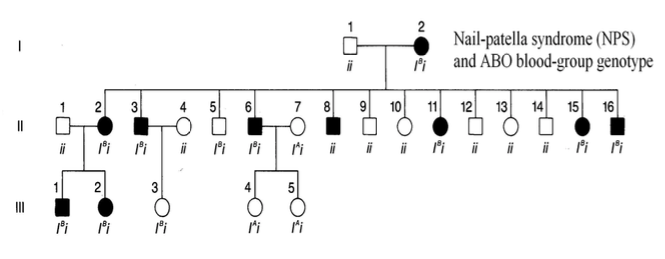
ここで、recombinantとnon-recombinantの数はいくつでしょうか?
答えは
- recombinant = 3(IIの5, 8とIIIの3)
- non-recombinant = 15(IIの2, 3, 6, 9, 10, 11, 12, 13, 14, 15, 16とIIの1, 2, 4, 5)
となります。
よって、
- θ= 3/(3+15) = 0.167
となります。
この方法をdirect methodと呼ぶことがありますが、
- 浸透率100%を仮定している
- 欠損値はない(全員が協力してくれた)
など、かなり強い前提を置いています。このため、この前提が崩れるとバイアスが大きく出てしまうという問題点があります。
おまけ:Genetic MapとPhysical Map
cMやθ以外にも、bpやMbなどの表記があり、知識がごちゃごちゃしてしまいそうですが、これらの単位はgenetic mapとphysical mapという異なる地図なので単位が違うのです。非常に簡略化して表にすると、以下のようになります:
|
Genetic Map |
Physical Map |
|
cM |
bp |
となります。
また、genetic mapとphysical mapの関連性ですが、およそ1 cM = 0.5-1Mbと考えられています。繰り返しますが「およそ」の関連性ですので、完璧に一致すると考えないでください。
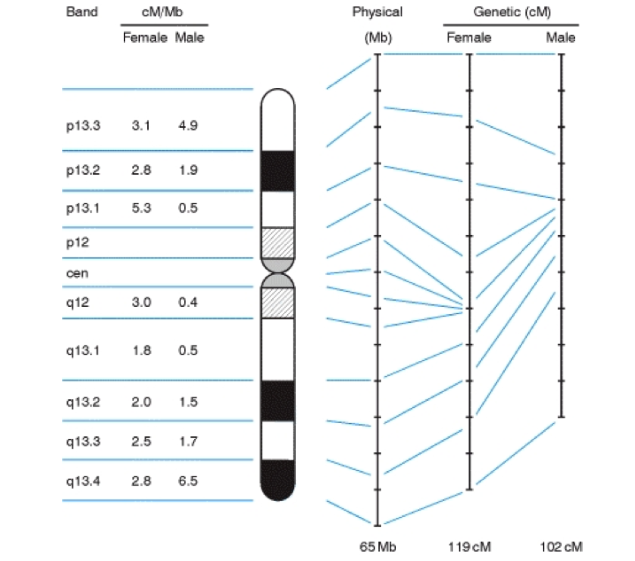
Physical MapとGenetic Mapを図示すると、上の図のようになります。
Genetic Mapには実は男女差があり、女性の方が若干長く出ています。
Linkage analysesについて
ここからはLinkage analysisについて解説していきます。
まずは以下の図を見てみましょう。
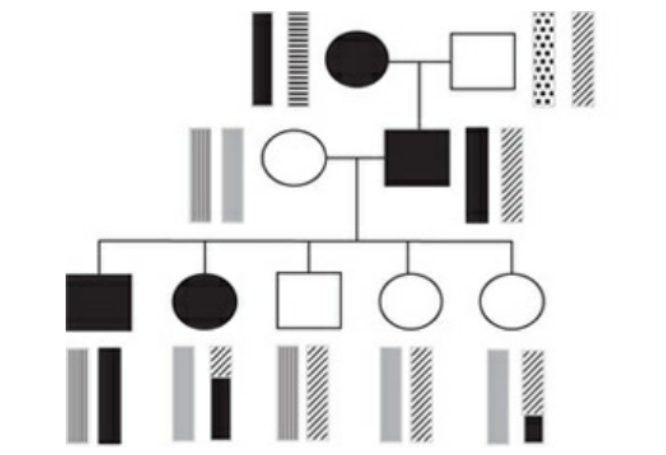
常染色体優性遺伝で、浸透率が100%であったとしましょう。
この家系の祖母から父へ、父から息子1人、娘1人に疾患が遺伝しています。
祖母から父への遺伝、父から第一子(息子)にはは原因遺伝子のある染色体の100%の情報が遺伝しているのがわかります。
一方で、父から娘(第二子)には、recombinationのため染色体の70%ほどの遺伝情報しか遺伝していませんが、それでも疾患を発症しています。
最後に、第5子には、recombinationのため、原因遺伝子のある染色体の30%ほどが父から伝達されていますが、疾患は発症していません。
Recombination fractionを利用する
このRecombinationの性質を利用して、疾患遺伝子の場所を推定することができます。
例えば、祖母と父の場合はrecombinationが起こっていないため、疾患の原因遺伝子の場所を染色体上から特定するのは困難です。
ですが、子供の世代ではrecombinationが起こっているため、ある程度の推定をすることができます。
例えば、第2子には持っていて、第5子には持っていない部位が疾患の原因遺伝子のある部位が候補となります。
もちろん、1つの家族のみで行うのではなく、複数の家族のデータをできるだけ集めてこの疾患の原因遺伝子が染色体上のどこにあるのかを推定していきます。
LOD scoreについて
LOD はlogarithm of oddsを意味し、
- odds for linkage: linkageを支持するodds
- odds against linkage: linkageを反対するodds
の比になります。Logはnatural logではなく、10を使用します(log10)。
この比はodds ratioでして、分子は計測されたデータのlinkageにおけるlikelihoodになるため、L(θ< ½)となります。
分母は、linkageがなかった場合の仮定になるため、θ=1/2になります。このため、L(0.5)となります。よって、ORは
- OR = L(θ< 0.5)/L(q = 0.5)
となります。ORの解釈ですが、
- OR > 1であれば、その家庭内でlinkageを示唆
- OR < 1であれば、linkageがないことを示唆
となります。
さらにORをLogにしたLODスコアは、2つの座位が強く連鎖しているサンプルから得られたデータらしいか、それとも純粋に偶然にそのようなデータが得られたらしいのか、の二つの比です。
正のLODスコアはlinkageの存在を示唆し、逆に負のLODスコアはこれらの座位がlinkageしていなさそうであることを示す。
LOD scoreの計算方法
LOD scoreの計算方法ですが、
- 家系図を作成する
- Recombinationの確率を用意する(0, 0.1, 0.2, 0.3, 0.4など)
- 用意した確率でLOD scoreを計算してみる
- 最も高いLOD scoreを与えた確率を、予測値として採用する
の4つの手順になります。
さらに、このORをlog10で対数かしたものをLOD scoreやZ-scoreと呼び、
- LOD(θ) = log10(L(q)/L(0.5)) = log10(OR) = Z(q)
となります。ここで、
- L(q) = (1–θ)NRx θR
- L(0.5) = 0.5 (NR + R)
となります(R = #recombinant, NR = #non-recombinant)。
これで求められた最も高いLOD scoreをrecombinantの確率として採用します。この計算をそれぞれの家系で行い、計測した家庭ごとのZ-scoreを足します。
- Z(θ) = ΣZi(θ) for i = 1, 2, …., n
このoverall Z-score(つまり、Z(θ) )が
- +3を超える場合には統計学的有意差をもってlinkageがある
- -2を下回る場合に、統計学的有意差をもってlinkageがない
ことを示唆します。
Genome Scanへの応用
LOD scoreはgenome scanへも応用されることがあります。例えば、
- Microsatellite
- SNP genome scan
などを利用して、同様にlinkageのlikelihoodを評価することができます。
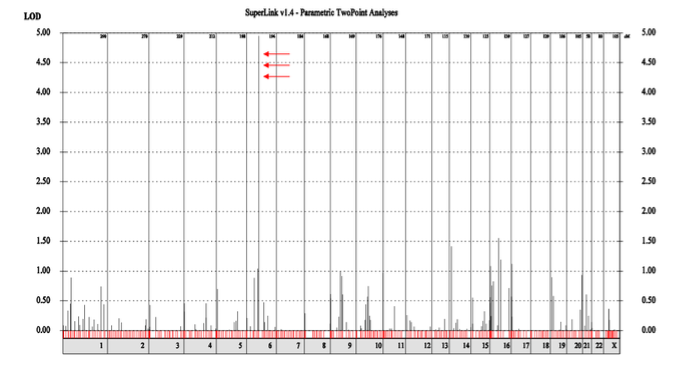
少し見辛いですが、上で説明した同様のプロセスをすべての染色体で行いLODを計算したものは上の図のようになります。
6番染色体のある部位でLODが非常に高くなっているのが分かります。
他のLinkage methodについて
Linkage 解析の他の手法ですが、LODを使用したもの以外にも、
- Non-parametric法
- Affected only analysis
- Allele sharing method
など、いくつかあります。
こちらに関しては、次回に説明できればと思います。